Epidemie
Stochastyczne i deterministyczne modele epidemii
Zjawiska opisywane na poziomie "mikro" przez procesy Markowa zachowują się na poziomie "makro" jak funkcje deterministyczne, będące rozwiązaniami równań różniczkowych. Można to zaobserwować na przykładzie prostego (ale, niestety, bardzo aktualnego w chwili pisania tego artykułu) modelu początkowej, wykładniczej fazy epidemii.
Rozważmy bardzo dużą populację. Niech 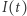 oznacza liczbę zarażonych w chwili
oznacza liczbę zarażonych w chwili 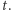 Zakładamy, że w krótkim okresie czasu
Zakładamy, że w krótkim okresie czasu 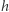 każdy z nich zaraża średnio
każdy z nich zaraża średnio 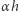 innych ludzi, ale przy tym z prawdopodobieństwem
innych ludzi, ale przy tym z prawdopodobieństwem 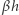 zdrowieje lub umiera i przestaje zarażać. Deterministyczny model jest następujący:
zdrowieje lub umiera i przestaje zarażać. Deterministyczny model jest następujący:
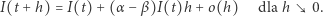 |
(1) |
(Traktujemy 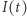 jako wielkość ciągłą. Symbol
jako wielkość ciągłą. Symbol 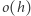 oznacza nieskończenie małą rzędu wyższego niż
oznacza nieskończenie małą rzędu wyższego niż 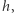 czyli funkcję spełniającą równość
czyli funkcję spełniającą równość 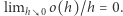 ) Oczywiście (1) sprowadza się do zwyczajnego równania różniczkowego:
) Oczywiście (1) sprowadza się do zwyczajnego równania różniczkowego:
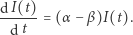 |
Popatrzmy na to samo zjawisko z bliska (w skali mikro). Potraktujmy 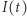 jako zmienną losową o wartościach całkowitoliczbowych. Ewolucja procesu
jako zmienną losową o wartościach całkowitoliczbowych. Ewolucja procesu 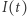 jest opisana równaniami
jest opisana równaniami
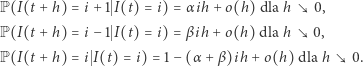 |
(2) |
Zakładamy, że 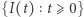 jest procesem Markowa, to znaczy ewolucja procesu po chwili
jest procesem Markowa, to znaczy ewolucja procesu po chwili 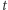 zależy tylko od
zależy tylko od 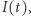 czyli stanu w chwili
czyli stanu w chwili 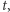 a nie od wcześniejszej "historii" procesu. Jest to skokowy proces Markowa: w losowych momentach
a nie od wcześniejszej "historii" procesu. Jest to skokowy proces Markowa: w losowych momentach 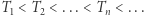 proces rośnie lub maleje o 1. Żeby lepiej zrozumieć strukturę tego procesu i sposób jego symulowania, przyjrzyjmy się czasom pomiędzy skokami,
proces rośnie lub maleje o 1. Żeby lepiej zrozumieć strukturę tego procesu i sposób jego symulowania, przyjrzyjmy się czasom pomiędzy skokami, 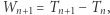 gdzie
gdzie
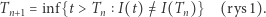 |
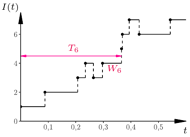
Rys. 1
Okazuje się (co zostanie uzasadnione na końcu artykułu), że 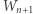 jest zmienną losową o rozkładzie wykładniczym z parametrem
jest zmienną losową o rozkładzie wykładniczym z parametrem 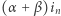 . Ponadto
. Ponadto
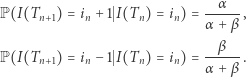
Łatwo w to uwierzyć, patrząc na równania (2).
Przebieg procesu 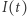 symulujemy zatem, losując kolejno czasy
symulujemy zatem, losując kolejno czasy 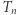 i stany
i stany 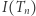 zgodnie z dwiema powyższymi regułami. Chemicy, biolodzy i epidemiolodzy znają tę metodę pod nazwą algorytmu Gillespie'go. W rzeczywistości taki opis skokowego procesu Markowa podał znakomity amerykański matematyk czeskiego pochodzenia, Joseph Doob. Przykładowy przebieg krótkich symulacji przedstawiamy w poniższej tabelce i na rysunku 1.
zgodnie z dwiema powyższymi regułami. Chemicy, biolodzy i epidemiolodzy znają tę metodę pod nazwą algorytmu Gillespie'go. W rzeczywistości taki opis skokowego procesu Markowa podał znakomity amerykański matematyk czeskiego pochodzenia, Joseph Doob. Przykładowy przebieg krótkich symulacji przedstawiamy w poniższej tabelce i na rysunku 1.
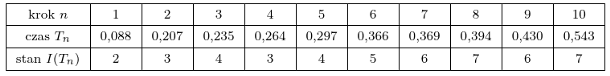
Rys. 2
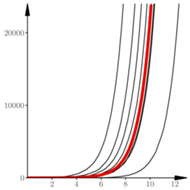
Rys. 3 Skala logarytmiczna
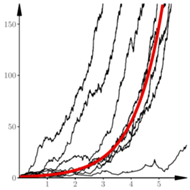
Rys. 4 Początkowe fragmenty 20 trajektorii
Popatrzmy teraz na dłuższe przebiegi symulacji i porównajmy trajektorie procesu losowego z rozwiązaniem równania różniczkowego, czyli funkcją wykładniczą 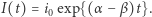 Przyjęliśmy następujące wartości parametrów:
Przyjęliśmy następujące wartości parametrów: 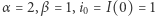 (śledzimy rozwój epidemii od pierwszego zarażonego). Na rysunku 2 przedstawionych jest 20 niezależnych trajektorii procesu Markowa. Spośród tych 20 trajektorii 12 wpadło dość wcześnie w stan 0. Jest to stan "pochłaniający": jeśli
(śledzimy rozwój epidemii od pierwszego zarażonego). Na rysunku 2 przedstawionych jest 20 niezależnych trajektorii procesu Markowa. Spośród tych 20 trajektorii 12 wpadło dość wcześnie w stan 0. Jest to stan "pochłaniający": jeśli 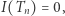 to dla każdego
to dla każdego 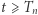 mamy
mamy 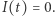 Pogrubiona, kolorowa linia to wykres funkcji wykładniczej.
Pogrubiona, kolorowa linia to wykres funkcji wykładniczej.
Uderzającą cechą procesu losowego opisanego równaniami (2) jest zgodność trajektorii w makroskali z rozwiązaniem równania różniczkowego, czyli z funkcjami wykładniczymi 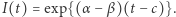 Poszczególne realizacje procesu różnią się jednak wyraźnie przesunięciem w fazie
Poszczególne realizacje procesu różnią się jednak wyraźnie przesunięciem w fazie  To widać na rysunku 3, na którym oś pionowa została przedstawiona w skali logarytmicznej. Rysunek 4 pokazuje te same 20 trajektorii w początkowym okresie rozwoju. Wspomniane wyżej przesunięcia w fazie wynikają z losowych fluktuacji na początku. Rezultatem tych losowych fluktuacji jest to, że 12 spośród przedstawionych trajektorii jest "pochłoniętych" przez zero. Dalszy przebieg procesu jest niemal deterministyczny.
To widać na rysunku 3, na którym oś pionowa została przedstawiona w skali logarytmicznej. Rysunek 4 pokazuje te same 20 trajektorii w początkowym okresie rozwoju. Wspomniane wyżej przesunięcia w fazie wynikają z losowych fluktuacji na początku. Rezultatem tych losowych fluktuacji jest to, że 12 spośród przedstawionych trajektorii jest "pochłoniętych" przez zero. Dalszy przebieg procesu jest niemal deterministyczny.
Model SIR
Rozważmy populację złożoną z 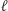 osobników. Niech
osobników. Niech 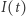 oznacza liczbę zarażonych w chwili
oznacza liczbę zarażonych w chwili 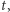 zaś
zaś 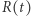 łączną liczbę uodpornionych i zmarłych. Liczba osobników narażonych na zakażenie jest równa
łączną liczbę uodpornionych i zmarłych. Liczba osobników narażonych na zakażenie jest równa 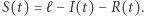 Osobnik typu
Osobnik typu 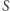 może przejść do kategorii
może przejść do kategorii 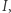 a stąd do kategorii
a stąd do kategorii 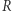 (stąd nazwa "model SIR"). Schematycznie:
(stąd nazwa "model SIR"). Schematycznie:
 |
Zakładamy, że w krótkim okresie czasu 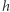 każdy osobnik
każdy osobnik 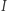 zaraża średnio
zaraża średnio 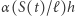 innych ludzi, a z prawdopodobieństwem
innych ludzi, a z prawdopodobieństwem 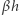 zdrowieje lub umiera i przestaje zarażać. Zauważmy, że jeśli
zdrowieje lub umiera i przestaje zarażać. Zauważmy, że jeśli 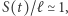 to dostajemy model fazy wykładniczej, przedstawiony w zadaniu poprzednim.
to dostajemy model fazy wykładniczej, przedstawiony w zadaniu poprzednim.
Klasyczny model deterministyczny jest następujący:
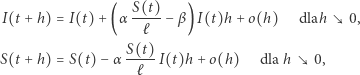 |
(3) |
oraz 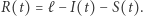 Oczywiście (3) jest układem równań różniczkowych zwyczajnych:
Oczywiście (3) jest układem równań różniczkowych zwyczajnych:
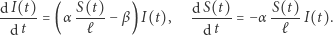 |
(4) |
W skali mikro traktujemy 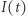 i
i 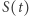 jako zmienne losowe o wartościach całkowitoliczbowych. Stan układu jest parą
jako zmienne losowe o wartościach całkowitoliczbowych. Stan układu jest parą 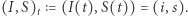 Ewolucja procesu jest opisana równaniami
Ewolucja procesu jest opisana równaniami
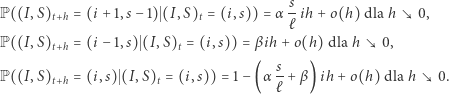 |
(5) |
Symulacja procesu opisanego powyższymi równaniami również może zostać przeprowadzona przy użyciu algorytmu Gillespie'go. Tym razem jednak musimy monitorować dwie wielkości - liczbę zainfekowanych 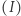 oraz liczbę osobników podatnych na zakażenie
oraz liczbę osobników podatnych na zakażenie 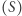 . Jeśli liczby te wynoszą
. Jeśli liczby te wynoszą 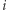 i
i 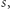 odpowiednio, to czas oczekiwania na kolejne "zdarzenie" (czyli zainfekowanie nowej osoby lub wyzdrowienie/śmierć osoby zarażonej) ma rozkład wykładniczy z parametrem
odpowiednio, to czas oczekiwania na kolejne "zdarzenie" (czyli zainfekowanie nowej osoby lub wyzdrowienie/śmierć osoby zarażonej) ma rozkład wykładniczy z parametrem 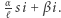 Kiedy już dojdzie do zdarzenia, to z prawdopodobieństwem
Kiedy już dojdzie do zdarzenia, to z prawdopodobieństwem 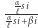 jest to nowe zarażenie (oraz wyzdrowienie/śmierć w przeciwnym przypadku). Nowe stany odpowiadające tym dwóm rodzajom zdarzeń to, odpowiednio,
jest to nowe zarażenie (oraz wyzdrowienie/śmierć w przeciwnym przypadku). Nowe stany odpowiadające tym dwóm rodzajom zdarzeń to, odpowiednio, 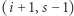 oraz
oraz 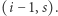 Oczywiście symulacja kończy się, kiedy
Oczywiście symulacja kończy się, kiedy 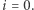
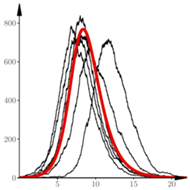
Rys. 5 Trajektorie procesu 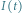 w modelu SIR
w modelu SIR
Na rysunku 5 widać kilka trajektorii procesu opisanego równaniami (5), wraz z zaznaczonym kolorem rozwiązaniem równania różniczkowego (4). Przyjęliśmy następujące wartości parametrów: 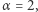
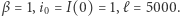

Rys. 6 Liczba wszystkich dotychczas zainfekowanych. Przerywaną linią oznaczono rozmiar populacji. We wszystkich symulowanych przebiegach po wygaśnięciu epidemii około 1000 osób pozostaje w stanie 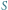 (są to osoby, które nie zostały zarażone)
(są to osoby, które nie zostały zarażone)
Początkowo trajektorie zachowują się tak, jak w modelu poprzednim: najpierw sporo losowych fluktuacji, później następuje faza wykładniczego wzrostu. Jeszcze później widać zupełnie inne zjawisko: hamowania wzrostu wskutek spadku liczby narażonych 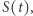 aż do zupełnego wygaśnięcia epidemii.
aż do zupełnego wygaśnięcia epidemii.
Dla różnych realizacji tego samego procesu SIR widać duże przesunięcia w fazie, wynikające z losowego charakteru początkowego fragmentu. Losowe fluktuacje (głównie początkowe) mają też pewien (niewielki) wpływ na maksymalną liczbę zarażonych (maksima poszczególnych krzywych).
W opracowaniach dotyczących rozwoju epidemii często przedstawia się również liczbę wszystkich osób, które zostały zainfekowane do danego momentu (tzn. liczbę zdarzeń polegających na zarażeniu nowej osoby). Liczba ta odpowiada procesowi 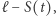 widocznemu na rysunku 6. Warto zwrócić uwagę na niewielkie fluktuacje liczby wszystkich osób, które zostały zakażone do czasu wygaśnięcia epidemii.
widocznemu na rysunku 6. Warto zwrócić uwagę na niewielkie fluktuacje liczby wszystkich osób, które zostały zakażone do czasu wygaśnięcia epidemii.
Przedstawione w tym artykule modele rozwoju epidemii są skrajnie uproszczone i nie nadają się do ilościowego opisu rzeczywistego zjawiska. Niemniej nawet takie modele pozwalają trochę zrozumieć mechanizm epidemii na poziomie jakościowym.
Dlaczego czasy oczekiwania na skok mają rozkład wykładniczy?
Aby udowodnić, że zmienna 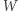 ma rozkład wykładniczy z parametrem
ma rozkład wykładniczy z parametrem 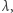 wystarczy pokazać, że zmienna
wystarczy pokazać, że zmienna 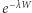 ma rozkład jednostajny na odcinku
ma rozkład jednostajny na odcinku ![|[0,1],](/math/temat/matematyka/zastosowania/2020/05/31/stochastyczne-i-deterministyczne-modele-epidemii/4x-cd66812e4336a9c30f1a502919ecb4a1cd00bec1-im-33,33,33-FF,FF,FF.gif) tzn. że dla dowolnego
tzn. że dla dowolnego 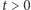 :
: 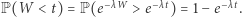
Przyjrzyjmy się czasowi pierwszego skoku, 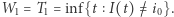 Nietrudno uwierzyć, że
Nietrudno uwierzyć, że
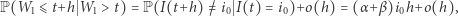 |
gdzie druga równość wynika z (2). Jeśli 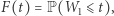 to dostajemy stąd równanie
to dostajemy stąd równanie 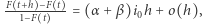 zatem
zatem 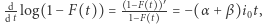 skąd wnioskujemy, że
skąd wnioskujemy, że 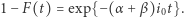 Identyczne rozumowanie stosuje się do
Identyczne rozumowanie stosuje się do 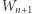 i prowadzi ono do analogicznego rezultatu, z
i prowadzi ono do analogicznego rezultatu, z 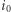 zastąpionym przez
zastąpionym przez 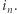
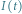 w modelu SIR
w modelu SIR (są to osoby, które nie zostały zarażone)
(są to osoby, które nie zostały zarażone)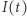 zmniejsza liczbę osób narażonych na zarażenie. To uproszczenie jest uzasadnione na początku epidemii.
zmniejsza liczbę osób narażonych na zarażenie. To uproszczenie jest uzasadnione na początku epidemii.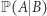 to prawdopodobieństwo zdarzenia
to prawdopodobieństwo zdarzenia 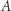 pod warunkiem zajścia zdarzenia
pod warunkiem zajścia zdarzenia 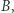 czyli
czyli 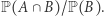
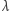 będzie dodatnią liczbą rzeczywistą. Jeśli wylosujemy liczbę
będzie dodatnią liczbą rzeczywistą. Jeśli wylosujemy liczbę 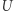 jednostajnie z odcinka
jednostajnie z odcinka ![|[0, 1]](/math/temat/matematyka/zastosowania/2020/05/31/stochastyczne-i-deterministyczne-modele-epidemii/3x-bdedb8ea18e9e3699690a6014f7e241821d2ef78-im-33,33,33-FF,FF,FF.gif) (tzn. szansa na to, że
(tzn. szansa na to, że 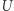 wpadnie w dowolnie wybrany odcinek
wpadnie w dowolnie wybrany odcinek ![|J⊂ [0,1],](/math/temat/matematyka/zastosowania/2020/05/31/stochastyczne-i-deterministyczne-modele-epidemii/5x-bdedb8ea18e9e3699690a6014f7e241821d2ef78-im-33,33,33-FF,FF,FF.gif) jest równa długości
jest równa długości 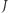 ), to zmienna losowa
), to zmienna losowa 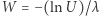 ma rozkład wykładniczy z parametrem
ma rozkład wykładniczy z parametrem