Epidemie
Pół szklanki mocnego kodu
Zanim dopadnie nas grypa
Czy lubicie długoterminowe prognozy pogody? Odbija się w nich głęboko zakorzeniona ludzka wiara, że odległą przyszłość można przewidzieć - na przekór efektowi motyla. No cóż, sam muszę przyznać, że cieszę się, gdy grudniowe prognozy przewidują piękną, słoneczną i mroźną aurę na zimowe ferie; ale jeśli zapowiadają ponury mokry standard, wtedy ratuję się myślą, że to przecież tylko prognoza...
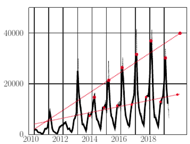
Średnia miesięczna liczba zachorowań na dzień (uśrednienie trochę wygładza wykres). Górna kolorowa prosta została dopasowana do pików z lat 2014-2018 metodą najmniejszych kwadratów. Analogicznie dopasowano dolną do chwil listopadowej stagnacji
Oczywiście można przewidywać nie tylko pogodę: na przykład zimą byłoby całkiem ciekawie przepowiedzieć rozwój corocznej epidemii grypy. I właśnie teraz, mimo braku wiedzy medycznej, uzbrojony jedynie w serię liczb, prosty model i kilka linii kodu (wybiorę moje ulubione środowisko obliczeniowe Octave) spróbuję przewidzieć, jak będzie przebiegać w tym sezonie.
Szczerze mówiąc, zdziwiłbym się, gdyby przyszłość posłuchała się moich prognoz: użyte przeze mnie narzędzia są raczej mało wyrafinowane. Ale... gdyby przewidywania się sprawdziły... czyż nie byłoby zabawnie?
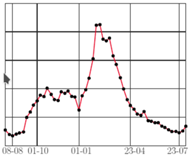
Skorzystamy z publicznie dostępnych danych NIZP-PZH na temat raportowanej przez lekarzy w całej Polsce liczby zachorowań na grypę (jak je zdobyć samemu, pisaliśmy w Delcie 10/2019). Zaczniemy od naszkicowania wykresu zachorowań w ostatnich latach, zobacz rysunek obok. (Dane zbierane są cztery razy w miesiącu).
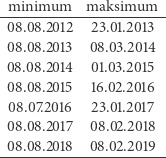
Wykres jest mało regularny, niemniej możemy zauważyć, że w pierwszym kwartale każdego roku ma silny pik, a w sierpniu - wyraźne minimum. Tabelka obok pokazuje daty, gdy zostało osiągnięte ekstremum, a dane do niej można wygenerować kodem:

W programie przyjęliśmy, że w zmiennej infected znajdują się liczby zachorowań, które wcześniej ściągnęliśmy z NIZP-PZH, a w days - daty odpowiadających pomiarów. Funkcja findpeaks z pakietu signal znajduje położenia loc lokalnych maksimów danych zawartych w wektorze infected. Dodatkowo wymagamy, by odległość między maksimami wynosiła co najmniej 24 pomiary, czyli 6 miesięcy (jasne, są przecież odległe z grubsza o rok). W trzeciej linii wydobywamy z danych tylko te odpowiadające znalezionym maksimom, które w kolejnej linii wypisujemy na ekranie.
Minimum zawsze przypada na drugi tydzień sierpnia. Maksimum nie zachowuje się aż tak regularnie, ale jak możemy obliczyć, średnia odległość między maksimami na przestrzeni ostatnich 6 lat wynosi około 368 dni - czyli z grubsza też jeden rok (co w końcu nie powinno nas aż tak dziwić...).
Rzuca się w oczy, że od kilku lat piki zgłaszanej liczby zachorowań systematycznie rosną (niemal liniowo). Nie będąc lekarzem, ale za to trochę wyobrażając sobie, jak działa sprawozdawczość, przypuszczałbym, że za tym wzrostem nie stoi realny wzrost liczby wszystkich zachorowań na grypę w Polsce - ale że zapewne jest on efektem coraz lepiej działającego systemu zbierania danych (coraz więcej lekarzy odsyła stosowne formularze?). Gdyby więc liniowy trend miał utrzymać się jeszcze w tym sezonie, łatwo będzie przewidzieć orientacyjną szczytową liczbę zgłoszeń:

Całą robotę - dopasowanie wykresu prostej do maksimów - wykonuje pierwsza linia powyższego kodu: funkcja polyfit wyznacza metodą najmniejszych kwadratów wielomian 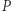 stopnia pierwszego, najmniej oddalony od punktów pomiarowych
stopnia pierwszego, najmniej oddalony od punktów pomiarowych 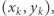 gdzie
gdzie 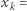 maxdays(k), a
maxdays(k), a 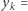 maxinfected(k). W drugiej linii obliczamy wartość tego wielomianu w dniu następnego maksimum zachorowań (przyjęliśmy, że wypadnie dokładnie za rok od ostatniego).
maxinfected(k). W drugiej linii obliczamy wartość tego wielomianu w dniu następnego maksimum zachorowań (przyjęliśmy, że wypadnie dokładnie za rok od ostatniego).
Typowy przebieg sezonu grypowego (tu dla sezonu 2015/2016)
Naturalnie w dłuższej perspektywie liniowy wzrost nie może się utrzymać, co zrozumiemy, przedłużając kolorowa prostą w lewo: "przewiduje" ona, że już w 2008 roku lekarze powinni byli zgłosić... ujemną liczbę zachorowań.
Modelowanie w wybranym sezonie
Powyższe miało charakter czysto rozrywkowy, więc czas na coś poważniejszego. Prawie każdy sezon chorobowy ma podobny przebieg (rysunek obok):
- dosyć szybki wzrost zachorowań od połowy sierpnia,
- z początkiem października następuje stabilizacja liczby zachorowań, a nawet - w pierwszym tygodniu stycznia - jej chwilowy spadek,
- od początku stycznia - kilkutygodniowa faza szybkiego wzrostu liczby zachorowań,
- osiągnięcie szczytowej liczby zachorowań (najczęściej w lutym),
- szybkie zmniejszanie się liczby zachorowań gdzieś tak do końca kwietnia,
- powolny spadek do kolejnego minimum.
Czy więc dałoby się - dysponując bieżącymi danymi ze strony internetowej NIZP-PZH - przewidzieć rozwój tegorocznej grypy? Do tego wykorzystamy następujący, bardzo prosty, model zachorowań. Niech 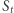 oznacza liczbę osób podatnych na wirusa (ale na razie zdrowych) w dniu
oznacza liczbę osób podatnych na wirusa (ale na razie zdrowych) w dniu 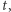 z kolei
z kolei 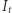 - liczbę osób zarażonych (i przy tym zarażających), a
- liczbę osób zarażonych (i przy tym zarażających), a 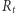 - liczbę osób, które już nie mogą być zarażone ani zarażać, bo np. wyzdrowiały i nabrały odporności. W modelu przyjmujemy następującą zależność
- liczbę osób, które już nie mogą być zarażone ani zarażać, bo np. wyzdrowiały i nabrały odporności. W modelu przyjmujemy następującą zależność 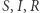 w dniu następnym od tychże w dniu obecnym:
w dniu następnym od tychże w dniu obecnym:

Gdybyśmy znali współczynniki modelu: 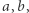 to moglibyśmy go użyć do wyznaczenia przyszłych wartości
to moglibyśmy go użyć do wyznaczenia przyszłych wartości 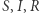 tak, jak w poniższej funkcji:
tak, jak w poniższej funkcji:

To bardzo zgrubny model, ale mamy nadzieję, że okaże się wystarczający, jeśli ograniczymy zakres jego stosowalności. Nie możemy liczyć na to, że uda się nam w modelu dopuszczającym tylko jeden pik zachorowań odtworzyć wieloletni przebieg choroby. Ale wymodelowanie okresu największej zachorowalności w sezonie, powiedzmy: od początku stycznia do końca kwietnia, wydaje się celem jak najbardziej realnym.
Ponieważ 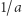 odpowiada średniej długości okresu, gdy zainfekowany zaraża, to możemy z dobrym skutkiem przyjąć, że
odpowiada średniej długości okresu, gdy zainfekowany zaraża, to możemy z dobrym skutkiem przyjąć, że 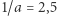 dnia (potem bierzemy zwolnienie i kładziemy się do łóżka), więc
dnia (potem bierzemy zwolnienie i kładziemy się do łóżka), więc 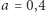 (na dzień).
(na dzień).
Nie znając dobrej wartości współczynnika 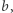 dopasujemy ją tak, by wartości
dopasujemy ją tak, by wartości  wyznaczone przez model jak najlepiej przybliżały te, które już znamy z raportów NIZP-PZH.
wyznaczone przez model jak najlepiej przybliżały te, które już znamy z raportów NIZP-PZH.
W tym celu definiujemy funkcję
![b ( F(b) = [I1(b),I2(b),...,In(b)],](/math/temat/matematyka/zastosowania/2019/11/25/Zanim_dopadnie_nas_grypa/1x-b8fb565a16de78fc88f94d07159ace56cd5291be-dm-33,33,33-FF,FF,FF.gif)
która dla zadanego 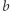 wyznacza (za pomocą powyższego modelu) wartości
wyznacza (za pomocą powyższego modelu) wartości 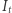 w punktach pomiarów w zadanym przez nas okresie. Następnie, korzystając z funkcji leasqr z pakietu optim, znajdziemy wartość
w punktach pomiarów w zadanym przez nas okresie. Następnie, korzystając z funkcji leasqr z pakietu optim, znajdziemy wartość 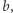 która zminimalizuje sumę kwadratów odchyleń wartości przewidywanych od wartości zmierzonych:
która zminimalizuje sumę kwadratów odchyleń wartości przewidywanych od wartości zmierzonych:

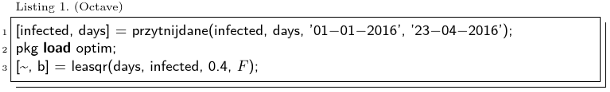
Najpierw ograniczamy zbiór danych infected, days do interesującego nas okresu (implementację funkcji przytnijdane można sobie z grubsza wyobrazić), a po załadowaniu pakietu optim funkcja leasqr wyznacza 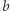 minimalizujące sumę kwadratów odchyleń (czwarty argument,
minimalizujące sumę kwadratów odchyleń (czwarty argument,  jest potrzebny, bo ta funkcja wyznacza wartości
jest potrzebny, bo ta funkcja wyznacza wartości 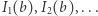 potrzebne do obliczenia odchyleń) - omówienie jej implementacji, wykorzystującej KMcK, pominiemy.
potrzebne do obliczenia odchyleń) - omówienie jej implementacji, wykorzystującej KMcK, pominiemy.
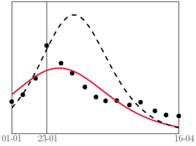
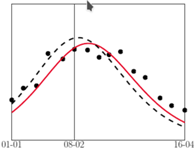
Jak widać na wykresach powyżej, dla tak wyznaczonego 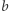 dostaliśmy kolorową krzywą, całkiem dobrze wpasowującą się w punkty pomiarowe (sezon 2016/2017 jest - jak widzieliśmy wcześniej - pod wieloma względami wyjątkowy i model nie przewiduje prawidłowo wielkości piku).
dostaliśmy kolorową krzywą, całkiem dobrze wpasowującą się w punkty pomiarowe (sezon 2016/2017 jest - jak widzieliśmy wcześniej - pod wieloma względami wyjątkowy i model nie przewiduje prawidłowo wielkości piku).
Okazuje się, że dla większości sezonów dostajemy podobną wartość optymalnego 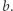 Aby więc przewidywać przyszłe epidemie, możemy po prostu skorzystać ze "średniej" wartości
Aby więc przewidywać przyszłe epidemie, możemy po prostu skorzystać ze "średniej" wartości 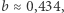 co zamyka sprawę (po prostu startujemy symulację z wartości znanych w konkretnym dniu). Z drugiej strony wybór uśrednionego
co zamyka sprawę (po prostu startujemy symulację z wartości znanych w konkretnym dniu). Z drugiej strony wybór uśrednionego 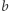 powoduje, że nie możemy reagować na ewentualne atypowości bieżącego sezonu (np. niesprzyjającą aurę lub szczególnie zjadliwy szczep wirusa, co w każdym przypadku powoduje zmianę współczynnika
powoduje, że nie możemy reagować na ewentualne atypowości bieżącego sezonu (np. niesprzyjającą aurę lub szczególnie zjadliwy szczep wirusa, co w każdym przypadku powoduje zmianę współczynnika 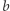 ). Wówczas lepiej dopasować nowe
). Wówczas lepiej dopasować nowe 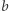 do bieżących danych. Przykładowy wynik dopasowania
do bieżących danych. Przykładowy wynik dopasowania 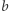 na podstawie jedynie czterech pomiarów ze stycznia (linia przerywana na wykresach obok) pokazuje, że możemy się wtedy sporo pomylić; jednak im więcej pomiarów wykorzystamy, tym lepsze będą przewidywania.
na podstawie jedynie czterech pomiarów ze stycznia (linia przerywana na wykresach obok) pokazuje, że możemy się wtedy sporo pomylić; jednak im więcej pomiarów wykorzystamy, tym lepsze będą przewidywania.
A zatem... zaglądając do mojej szklanej/krzemowej kuli...
przepowiadam, że:
przepowiadam, że:
- Następny sezon grypy... już się zaczął: w połowie sierpnia!
- Od października do końca grudnia raportowana liczba zachorowań nie przekroczy... 140 tysięcy w tygodniu! [Odczytany liniowy trend z pierwszego wykresu].
- Szczyt zachorowań przypadnie... w drugim tygodniu lutego! [Po prostu zakładam, że stanie się to za rok od ostatniego]. Będzie wtedy zgłaszanych aż 400 tysięcy zachorowań tygodniowo! [Z liniowego trendu na pierwszym wykresie; jednak nie wierzę w to zbytnio i mam nadzieję, że naprawdę będzie ich znacznie mniej... Chociaż, z drugiej strony, grypa jest chorobą zdradziecką, więc po poprzednim roku ciszy teraz może zaatakować ze zdwojoną siłą].
- Przebieg zachorowalności w I kwartale 2020 roku... będę na żywo monitorować i prognozować na mojej stronie internetowej już od teraz, co tydzień uwzględniając nowe dane! [Będę dopasowywać na bieżąco parametry modelu, minimalizując błąd średniokwadratowy, podobnie jak opisano powyżej].
No dobra, czas kończyć pisanie. Już w trakcie poczułem się - jakoś tak - niewyraźnie: boli mnie głowa, drapie w gardle i... tak jakby... łamie w kościach? Czyżby -
Aaa... apsik!...
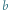 wyznaczonego na podstawie wszystkich danych. Czarna, przerywana linia: dla
wyznaczonego na podstawie wszystkich danych. Czarna, przerywana linia: dla 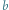 wyznaczonego na podstawie tylko pierwszych czterech pomiarów
wyznaczonego na podstawie tylko pierwszych czterech pomiarów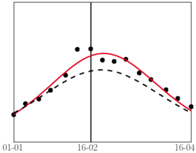
 zmienia się w czasie, podczas gdy my radośnie przyjmujemy, że jest stała.
zmienia się w czasie, podczas gdy my radośnie przyjmujemy, że jest stała.