Kącik biologiczny
Chytre sztuczki
Kary Mullis to zdecydowanie najbardziej kontrowersyjny z noblistów. Jako dziecko wysyłał żaby na wysokość 1,5 mili w skonstruowanych przez siebie rakietach. W 1968 roku, pod wpływem przemyśleń po zażyciu LSD, napisał artykuł „Kosmologiczne znaczenie odwrócenia czasu”, który opublikowało Nature.
Gdy pewnego letniego wieczoru wracał z pracy, wpadł mu do głowy pomysł, który zrewolucjonizował biologię molekularną.
Wymyślił prosty sposób na nieograniczone powielenie niemal dowolnego fragmentu DNA. Do tamtego czasu wymagało to prowadzenia hodowli bakteryjnych na dużą skalę, było bardzo pracochłonne i trwało przynajmniej dobę. Sposób Mullisa wymagał jednej probówki, 5 odczynników i w ciągu najwyżej kilku godzin dostarczał ogromnych ilości materiału.
Dwie nici w cząsteczce DNA biegną antyrównolegle i końce każdej z nich nie są równocenne. Oznaczamy je 3’ i 5’ – od numerów atomów węgla, do których mogą przyłączać się kolejne nukleotydy. Polimeraza DNA dołącza nowe nukleotydy według zasady komplementarności, wykorzystując jako matrycę rozplecioną na pojedyncze nici macierzystą cząsteczkę DNA. Jednak może ona dołączać nowe cegiełki tylko do nukleotydów, które mają wolne końce 3’. Nie może rozpocząć syntezy „od zera”, wymaga obecności tzw. startera – cząsteczki kwasu nukleinowego, która dostarczy pierwszego nukleotydu z wolnym końcem 3’. Dzięki temu lokalizacja starterów decyduje o miejscu startu syntezy nowej nici DNA.
Spróbujmy prześledzić następujący cykl reakcji:
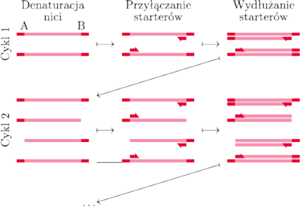
Chcemy namnożyć fragment DNA człowieka – odcinek AB. W pierwszym
kroku musimy rozdzielić dwuniciową cząsteczkę – co jest proste, gdyż
wystarczy ją podgrzać do 95
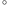 C. W ten sposób uzyskamy dwie
jednoniciowe matryce dla polimerazy. Do rozpoczęcia syntezy potrzebujemy
jeszcze dwóch starterów, dołączonych do matrycy dokładnie w punktach
A (starter „prawy”) i B (starter „lewy”).
C. W ten sposób uzyskamy dwie
jednoniciowe matryce dla polimerazy. Do rozpoczęcia syntezy potrzebujemy
jeszcze dwóch starterów, dołączonych do matrycy dokładnie w punktach
A (starter „prawy”) i B (starter „lewy”).
Starterami w naszej reakcji są krótkie, jednoniciowe fragmenty DNA, których syntezą zajmują się komercyjne firmy. Cetus Corporation, dla której pracował Mullis w latach 70. ubiegłego wieku, zajmowała się właśnie ich produkcją.
Zaprojektowanie i zsyntetyzowanie odpowiednich starterów wymaga znajomości sekwencji końców odcinka AB. Gdyby naszym materiałem wyjściowym był genom człowieka, takie startery musiałyby wiązać się z matrycowym DNA tylko w regionie AB wśród 3 miliardów par zasad.
Zakładając, że sekwencja nukleotydów w nici matrycowej jest losowa (każdy
z 4 nukleotydów ma jednakową szansę znalezienia się w dowolnym miejscu
sekwencji), szansa znalezienia dowolnej sekwencji o długości
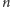 w genomie wynosi
w genomie wynosi
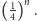 Aby starter był unikalny,
Aby starter był unikalny,
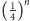 musi być
mniejsze niż
musi być
mniejsze niż
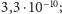 warunek ten jest spełniony dla
warunek ten jest spełniony dla
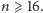 Zwykle stosuje się startery o długości 17–26 nukleotydów.
Zwykle stosuje się startery o długości 17–26 nukleotydów.
Kiedy więc do jednoniciowych matryc dodamy nieco starterów, polimerazy, wolnych nukleotydów i obniżymy trochę temperaturę, by startery mogły przyłączyć się do matrycy, polimeraza rozpocznie syntezę nowych nici. Jeśli powtórzymy ten cykl zmian temperatury, powiedzmy 30 razy, to po trzecim cyklu liczba cząsteczek potomnych – odcinków AB – zacznie rosnąć w postępie geometrycznym!
Tak właśnie działa PCR, czyli wymyślona przez Karego Mullisa łańcuchowa reakcja polimerazy. Nie wymaga uprzedniego wyizolowania odcinka AB z genomu – po prostu selektywnie go powiela. Nie wymaga dużej ilości materiału wyjściowego – wystarczy tylko jedna cząsteczka DNA matrycowego. Nie wymaga skomplikowanego sprzętu – współczesne maszyny do PCR to po prostu bloki grzejno-chłodzące, których temperaturą steruje procesor. Jeden cykl reakcji trwa przeciętnie minutę. Jedynym poważnym problemem związanym ze stosowaniem PCR jest jego czułość: każde zanieczyszczenie próbki (np. nasz własny DNA) namnoży się równie dobrze jak badany DNA, jeśli tylko startery przyłączą się do cząsteczki matrycowej. PCR wymaga dobrej techniki laboratoryjnej i stosowania wielu kontroli w każdej reakcji. Oczywiście, musimy też znać sekwencję, którą chcemy namnożyć.
Dzięki PCR można zidentyfikować przestępcę na podstawie DNA wyizolowanego ze śliny, którą ten zostawił na polizanym znaczku listu z pogróżkami. Można zdiagnozować chorobę genetyczną zarodka, pobierając zaledwie jedną jego komórkę. Można też zbadać DNA neandertalczyka, zachowany przez 30 tys. lat w skamieniałych kościach.
Bez wielkiej przesady można powiedzieć, że wynalazek Karego Mullisa zmienił biologię molekularną. Dla Cetus Corporation miał też wymiar bardziej bezpośredni – Hoffman-La Roche odkupił od niej patent na PCR za 300 milionów dolarów, co do dziś pozostaje rekordową sumą zapłaconą za patent w historii.