Zobaczyć niewidoczne
Każdy z nas może z łatwością wymienić zawody, których wykonywanie naraża ludzi na ciągły stres. Często stres jest związany z tym, że decyzje podejmowane w codziennej pracy wpływają na zdrowie (i życie) innych. Strażak, ratownik medyczny, chirurg, pilot, radiolog... Wszyscy muszą działać szybko, a koszt potencjalnych pomyłek może być dramatycznie wysoki. Warto zauważyć, że proces podejmowania decyzji w praktyce polega na analizie różnych danych (w czasie rzeczywistym), np. w przypadku danych medycznych mogą to być różne rodzaje (modalności) obrazów, zawierające różne informacje o pacjencie. Zobaczmy, jak sztuczna inteligencja może ułatwić proces podejmowania takich decyzji.
Obraz mówi więcej niż tysiąc słów
Skupmy się na analizie obrazów medycznych wykorzystywanych w onkologii. Celem takiej analizy, która z reguły jest przeprowadzana przez radiologa (lub fizyka medycznego), jest dostrzeżenie pewnych regionów w obrazie, które mogą być wykorzystane do postawienia diagnozy lub oceny tego, jak pacjent reaguje na zastosowany rodzaj leczenia. Bardzo często wykonywany jest ręczny opis (obrys) zmiany nowotworowej (w 2D lub w 3D), a następnie wyznaczana jest jej powierzchnia lub objętość. Oczywiście to nie jedyna wielkość liczbowa, która może "opisywać" (kwantyfikować) zmianę nowotworową i która może zostać wyekstrahowana z sekwencji obrazów medycznych. Obraz mówi więcej niż tysiąc słów - musimy tylko (i aż) wiedzieć, czego i jak szukać.
Bardzo istotną kwestią w analizie obrazów medycznych jest zapewnienie powtarzalności wyników. Wyobraźmy sobie sytuację, w której ten sam obraz jest ręcznie segmentowany przez dwie doświadczone osoby, z których pierwsza oznacza zmianę nowotworową w sposób bardziej "konserwatywny" (tj. stara się jak najdokładniej oznaczyć granicę nowotworu), a druga z osób oznacza nowotwór mniej dokładnie. Ten sam obraz, ten sam nowotwór, a wyniki liczbowe wyraźnie się różnią…Dodajmy do tego jeszcze fakt, że pacjent, gdy przychodzi do szpitala (załóżmy w czasie 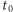 ), jest poddawany badaniu obrazowemu. Następnie jest leczony, a kolejne badania obrazowe są przeprowadzane w czasie
), jest poddawany badaniu obrazowemu. Następnie jest leczony, a kolejne badania obrazowe są przeprowadzane w czasie 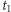 i
i 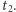 Możemy sobie łatwo wyobrazić, że jeśli poszczególne badania będą opisywane przez różne osoby, a przyjęte strategie segmentacji będą inne, to ocena progresu (lub regresu) choroby w czasie od
Możemy sobie łatwo wyobrazić, że jeśli poszczególne badania będą opisywane przez różne osoby, a przyjęte strategie segmentacji będą inne, to ocena progresu (lub regresu) choroby w czasie od 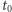 do
do 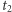 (np. wspomniana objętość nowotworu) może być niemiarodajna lub nawet myląca.
(np. wspomniana objętość nowotworu) może być niemiarodajna lub nawet myląca.
Na pomoc radiologom
Odpowiedzią na problem opisany powyżej jest automatyzacja wstępnej analizy (segmentacji) obrazów. Oczywiście nie dążymy do zastąpienia pracy radiologa, ale do jej ułatwienia, przyspieszenia i do zapewnienia takich warunków, by segmentacja była zawsze wykonywana w ten sam (powtarzalny) sposób. Do segmentacji możemy użyć zarówno tradycyjnych algorytmów opartych o techniki analizy obrazów (przykład kolejnych kroków algorytmu segmentacji mózgu w obrazie rezonansu magnetycznego głowy jest przedstawiony na rysunku 1), jak i metod wykorzystujących uczenie maszynowe (ang. machine learning). Drugą grupę algorytmów możemy podzielić na "tradycyjne" techniki uczenia maszynowego (tj. takie, które wykorzystują ręcznie wyekstrahowane cechy) i uczenie głębokie (ang. deep learning), w którym cechy ekstrahowane są automatycznie.
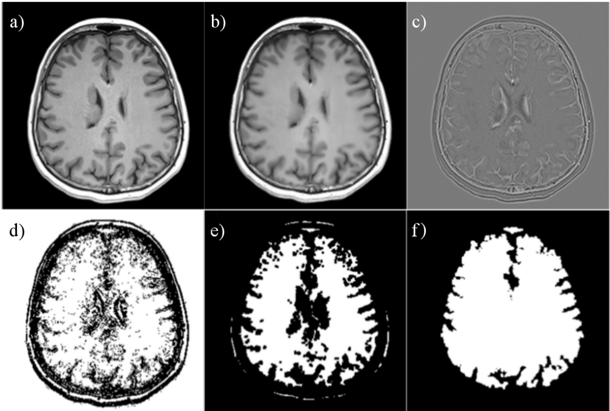
Rys. 1. Kolejne kroki przetwarzania obrazu wejściowego rezonansu magnetycznego (a) w przykładowym algorytmie segmentacji mózgu, opartym o techniki przetwarzania i analizy obrazów: (b) wynik dyfuzji anizotropicznej, (c) wynik filtracji obrazu (filtr Laplacian of Gaussian), (d) wyekstrahowane "granice" anatomiczne, (e) wynik erozji, (f) wysegmentowany region ( "maska") mózgu. Przykład został zaczerpnięty z publikacji [1].
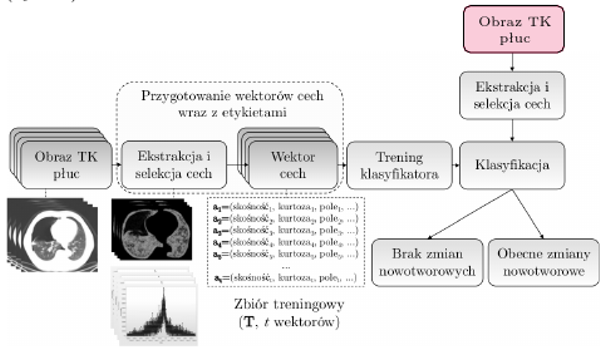
Tego rodzaju algorytmy są stosowane do klasyfikacji pikseli (lub tzw. wokseli w przypadku trójwymiarowym) do jednej z klas. Jeśli rozważymy problem klasyfikacji dwuklasowej (nazywanej też binarną), to pikselowi przypisywana jest etykieta jednej z dwóch klas (np. zmiana nowotworowa lub zdrowa tkanka). Każdy z pikseli jest opisany przez tzw. wektor cech, a przykłady pikseli należących do obu klas tworzą zbiór treningowy, który jest użyty w czasie treningu klasyfikatora (w procesie tzw. treningu nadzorowanego lub treningu z nauczycielem - patrz rysunek 2).
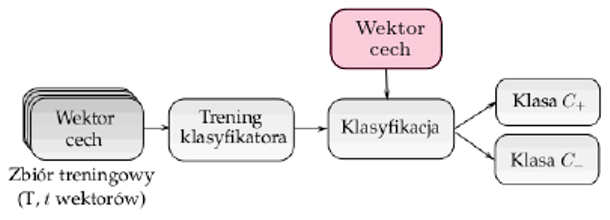
Rys. 2. Klasyfikacja nadzorowana - klasyfikator (przypisujący pikselom etykietę klasy) jest trenowany przy użyciu zbioru treningowego 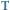 zawierającego
zawierającego  przykładów i jest wykorzystany do klasyfikacji nowych danych (tj. takich, które nie były użyte w czasie treningu) - na rysunku oznaczonych kolorem.
przykładów i jest wykorzystany do klasyfikacji nowych danych (tj. takich, które nie były użyte w czasie treningu) - na rysunku oznaczonych kolorem.
Łatwo zauważyć, że w przypadku cech ręcznie ekstrahowanych musimy je wcześniej zdefiniować. Żeby zdefiniować dobre cechy (czyli takie, które pozwolą na wytrenowanie wysokiej jakości klasyfikatora), musimy - ponownie - wiedzieć "czego szukamy". Intensywność (jasność) pikseli, cechy teksturalne, cechy wyekstrahowane po filtracji obrazu… To tylko nieliczne przykłady cech, które mogą opisywać obiekty w obrazie (rys. 3).
Rys. 3. Ręczna ekstrakcja cech opisujących obiekty jest zadaniem bardzo trudnym. Jakie cechy? Ile cech? Odpowiedź na te pytania musimy znaleźć przed treningiem naszego klasyfikatora.
Liczba cech może być bardzo duża, dlatego często wykonuje się dodatkową selekcję cech - wybieramy tylko najważniejsze cechy (tutaj z pomocą mogą przyjść np. algorytmy ewolucyjne).
W ostatnich latach w literaturze pojawia się coraz więcej technik opartych o automatyczną ekstrakcję cech. Bardzo dobrym przykładem takich automatycznych ekstraktorów są głębokie sieci neuronowe (ang. deep neural networks). Zauważmy, że głęboką sieć splotową można podzielić na dwie "części" - ekstraktor cech (zawierający np. warstwy splotowe, ang. convolutional layers, czy warstwy tzw. grupowania cech, ang. pooling) oraz klasyfikator. Co możemy zyskać, używając takich automatycznych ekstraktorów cech.
Zobaczyć (i zrozumieć) więcej
Możemy (przynajmniej spróbować) "zobaczyć" to, co niewidoczne gołym okiem. Jeśli mamy wystarczająco duży zbiór (zróżnicowanych) danych treningowych (co w praktyce jest, niestety, rzadkie), to możemy liczyć na to, że po treningu uzyskamy ekstraktor cech, o których moglibyśmy wcześniej nie pomyśleć. Spójrzmy na rysunek 4 - przedstawiamy na nim przykłady filtrów w sieci splotowej (której część ekstrahująca cechy składa się z jednej dwu- i jednej trójwymiarowej warstwy splotowej). Jak widać, część z przetworzonych przez te wytrenowane filtry obrazów przypomina takie obrazy, które moglibyśmy uzyskać, stosując różne (znane) algorytmy filtracji obrazów. Ale nie wszystkie… Konia z rzędem temu, kto wpadłby na zastosowanie właśnie takich filtrów do ekstrakcji cech!

Rys. 4. Wytrenowane filtry w warstwach splotowych (2D i 3D) mogą pozwolić na ekstrakcję wcześniej nieznanych cech z obrazów. Na rysunku widzimy przykłady przetworzonych obrazów. Czy te obrazy przypominają Ci takie, które moglibyśmy otrzymać, stosując standardowe techniki przetwarzania (filtracji) obrazów? Ciemniejszym kolorem zaznaczono część nowotworu, która została poprawnie wysegmentowana przy użyciu sieci głębokiej, a jaśniejszym kolorem zaznaczyliśmy fałszywe negatywy, tj. piksele, które zostały oznaczone przez sieć jako przedstawiające tkankę zdrową, a w rzeczywistości są częścią nowotworu. Ten rysunek został zainspirowany pracą [2].
Powyższa sieć (z dołączonymi warstwami klasyfikacyjnymi) została wykorzystana do segmentacji obrazów płuc otrzymanych za pomocą tomografii komputerowej (TK). W obrazach wyszukiwaliśmy zmiany charakteryzujące się tzw. wysokim wychwytem znacznika (substancji promieniotwórczej), wprowadzonego do ciała skanowanego pacjenta (ang. high-uptake lesions). Co ciekawe, do analizy tego rodzaju zmian wykorzystuje się tzw. pozytonową tomografię emisyjną (ang. positron emission tomography, PET) - w innym obrazowaniu, na przykład właśnie w TK, takie nowotwory są po prostu… niewidoczne. Spójrzmy na przykład pokazany na rysunku 5 - charakterystyka nowotworu jest bardzo trudna (lub niemożliwa) do "uchwycenia" w obrazie TK, bez wykorzystania obrazu PET.
Wyniki badań, które zaprezentowaliśmy w pracy [2], okazały się bardzo obiecujące. Głęboka sieć splotowa, wytrenowana przy użyciu obrazów TK, umożliwiła segmentację zmian o wysokim wychwycie w TK (jakość segmentacji zależała od wielkości nowotworu - im większy nowotwór, tym z większą pewnością był segmentowany). Zobaczenie "niewidocznego" (nie tylko w TK, ale też w innych modalnościach) może pomóc w jak najlepszym dostosowaniu rodzaju leczenia (i polepszeniu jakości życia) u pacjentów z chorobami nowotworowymi. Wykorzystajmy to, co widać, i to, co "ukryte" w obrazach medycznych, w efektywnej walce z rakiem.
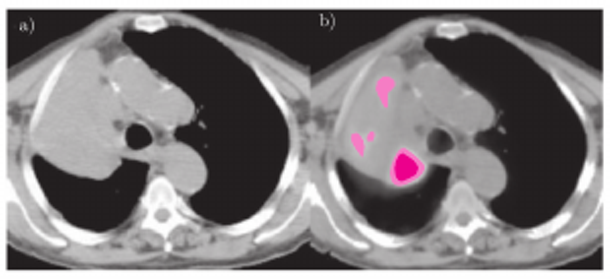
Rys. 5. Po nałożeniu obrazu PET na obraz TK (b) widać wyraźnie charakterystykę zmian nowotworowych, które nie są widoczne w obrazie TK (a). Przykład został zaczerpnięty z pracy [3].
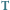 zawierającego
zawierającego  przykładów i jest wykorzystany do klasyfikacji nowych danych (tj. takich, które nie były użyte w czasie treningu) - na rysunku oznaczonych kolorem.
przykładów i jest wykorzystany do klasyfikacji nowych danych (tj. takich, które nie były użyte w czasie treningu) - na rysunku oznaczonych kolorem.